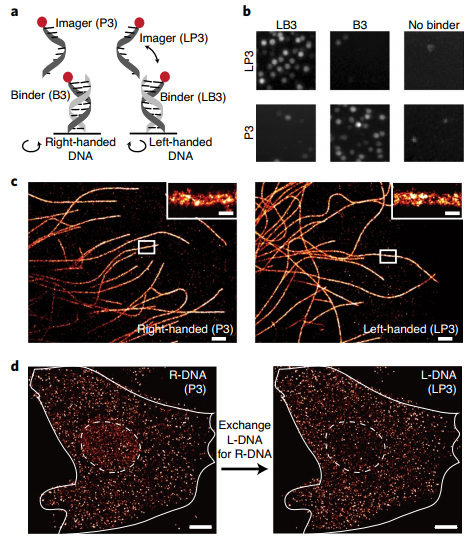
超分辨率显微镜逐渐成为生物学研究中有力的工具,但是获取大量不同目标样品的多路图像仍然具有挑战性。在这种情况下,DNA-PAINT技术(DNA point accumulation in nanoscale topography)【1】应运而生。DNA-PAINT技术是指通过短荧光标记的寡核苷酸用于纳米级定位成像的点积累短暂结合,可以在固定细胞内实现简单且易于实现的多路3D超分辨率成像并可以在体外实现纳米级别的空间分辨率【1】。但是DNA-PAINT技术存在一定的技术性限制,由于细胞核内的靶标与探针的DNA-DNA的杂交会造成信号背景较高。近日,来自德国柏林自由大学H. Ewers 研究组在Nature Biotechnology杂志上发表了一篇题为“Left-handed DNA-PAINT for improved super-resolution imaging in the nucleus”的文章,建立了新颖的左手螺旋DNA-PAINT技术(L-DNA-PAINT),对先前的DNA-PAINT技术进行了进一步的优化,大大提高了该技术的分辨率以及超分辨成像中的多路复用能力。超分辨率显微镜技术提高了人们对于细胞内结构细微之处的认识。单分子定位超分辨率显微镜(Single-molecule localization super-resolution microscopy,SMLM)通过每次几个分子的连续成像来实现对单分子纳米级分辨率的记录【2, 3】。DNA-PAINT技术是通过DNA杂交进行的,使用明亮的、光稳定的有机染料来获得最大的单分子定位分辨率。然而,任何的DNA分子在统计的平均值来说有22000个互补结合位点,这可能会导致DNA-PAINT技术的假阳性的出现。此外,转录组中也会出现不必要的DNA-RNA杂交。为了克服这一问题,作者们使用了左手螺旋的L-DNA来优化DNA-PAINT技术。L-DNA的物理化学性质与R-DNA基本相同,但是L-DNA在自然情况下不存在且不会与R-DNA之间杂交【3, 4】(图1a-b)。图1. L-DNA-PAINT与传统R-DNA-PAINT的比较首先,作者们对L-DNA-PAINT技术的表现进行了检验。作者们将HeLa细胞中的微管结构作为检验的平台,实验发现无论是R-DNA-PAINT还是L-DNA-PAINT技术都能够精准地表现出微管的中空结构(图1c),并且两者的分析分辨率均能够达到33-39nm左右。因此,作者们认为L-DNA-PAINT技术与R-DNA-PAINT技术在分辨率方面表现相似。
进一步地,作者们对两种技术对基因组DNA的杂交潜力进行检测。作者们发现,与R-DNA-PAINT技术相比,L-DNA-PAINT中细胞核中的定位降低到细胞质中的背景水平。另外,作者们对两种技术的假阳性情况进行了检测。通过BrdU(非天然核苷酸)整合到DNA复制位点的实验,作者们发现L-DNA-PAINT实验中可以对特定的复制中心进行精准标记。DNA-PAINT技术一个重要的亮点是可以对多个目标位点进行可视化研究。通过使用不同的L-DNA成像结合子(Imager-binder pairs)对PCNA以及Ki67进行染色,作者们发现两种分子都可以被染色,并且与先前报道的在细胞核内的定位类似【5】。总的来说,作者们所建立的优化版L-DNA-PAINT技术具有与传统R-DNA-PAINT相同的特异性、分辨率以及多路复用的能力,但R-DNA-PAINT技术相比具有更低的假阳性率以及更低的背景。未来,L-DNA-PAINT技术作为单分子定位超分辨率显微镜技术的重要实验方法能够更加精细地在纳米级别分辨率上可视化以及定量化DNA相关的分子,为进一步地对细胞内的结构进行解析提供了“更优解”。https://doi.org/10.1038/s41587-020-00753-y1. Jungmann,R. et al. Multiplexed 3D cellular super-resolution imaging with DNA-PAINT andExchange-PAINT. Nature methods 11, 313-318, doi:10.1038/nmeth.2835 (2014).2. Rust, M. J., Bates, M.& Zhuang, X. Sub-diffraction-limit imaging by stochastic opticalreconstruction microscopy (STORM). Nature methods 3, 793-795,doi:10.1038/nmeth929 (2006).3. Betzig, E. et al.Imaging intracellular fluorescent proteins at nanometer resolution. Science(New York, N.Y.) 313, 1642-1645, doi:10.1126/science.1127344 (2006).4. Hauser, N. C. et al.Utilising the left-helical conformation of L-DNA for analysing different markertypes on a single universal microarray platform. Nucleic acids research 34,5101-5111, doi:10.1093/nar/gkl671 (2006).5. Chagin, V. O. et al. 4DVisualization of replication foci in mammalian cells corresponding toindividual replicons. Nature communications 7, 11231, doi:10.1038/ncomms11231(2016).
文章来源:bioart